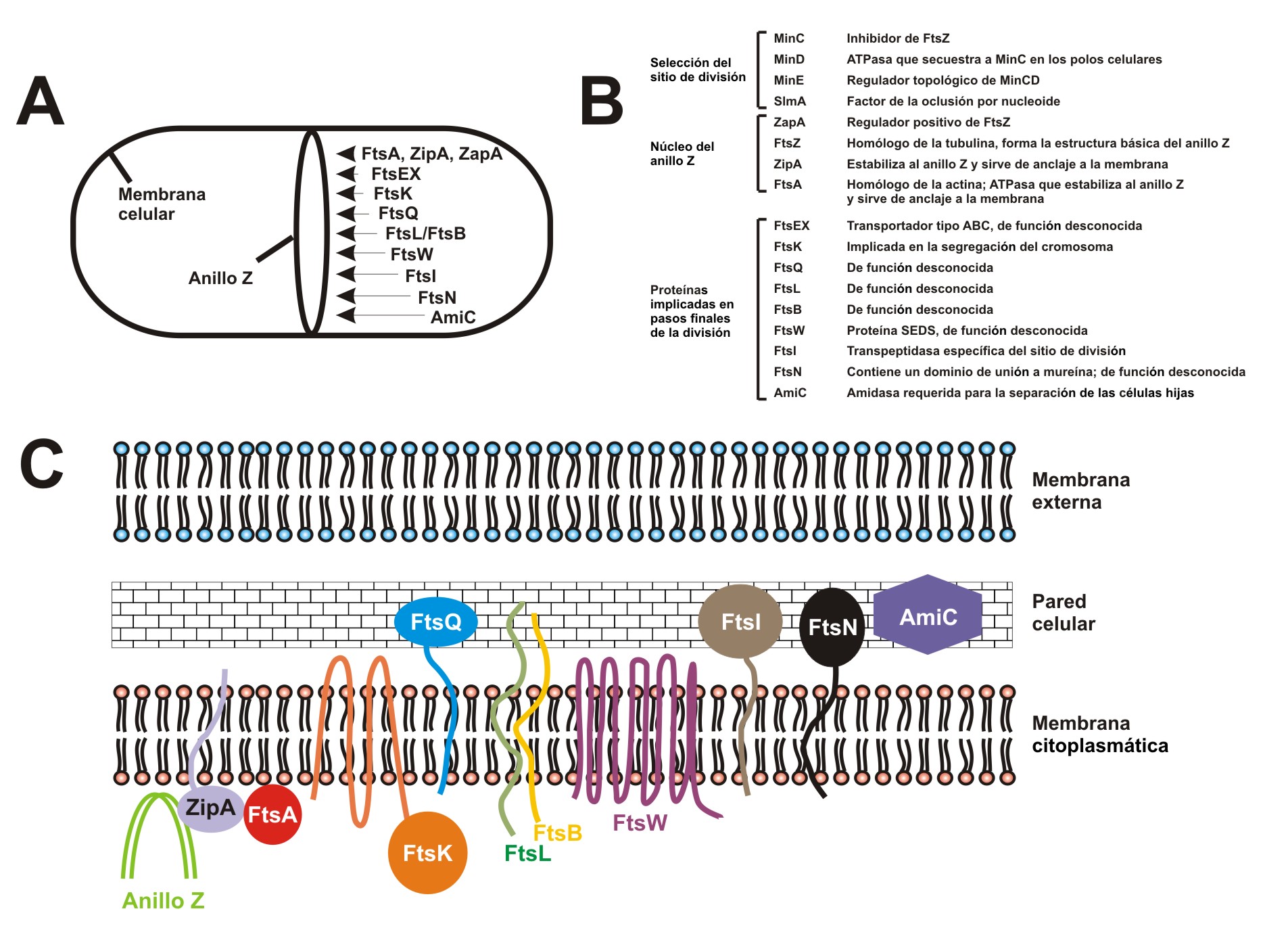
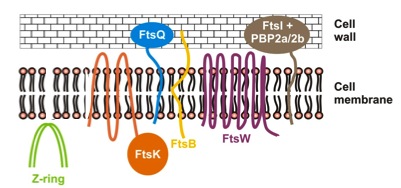
Una de nuestras líneas de investigación ha sido el estudio del proceso de división/elongación celular de Corynebacterium glutamicum debido a que tiene un modelo de división llamado división crepitante (snapping division) y posee una maquinaria de división «minimalista» cuando se compara con la maquinaria de Escherichia coli.
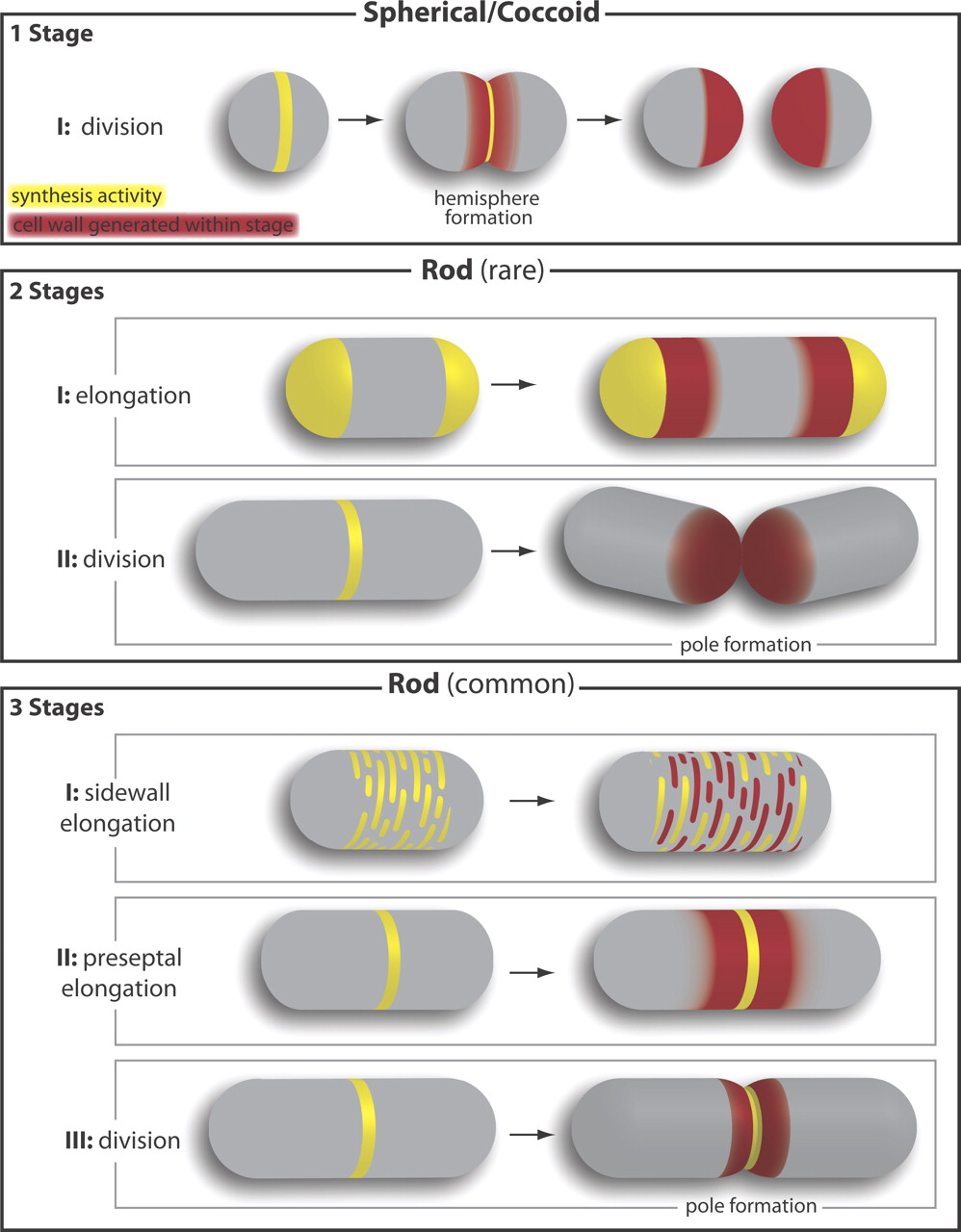
C. glutamicum muestra un modelo de crecimiento «raro» que los diferencia de los modelos clásicos de Escherichia coli, Bacillus subtilis o Streptococcus pneumoniae (Fig. 2). Los resultados obtenidos con C. glutamicum nos permitirán diseñar compuestos que inhiban la división celular en organismos patógenos relacionados como C. diphtheriae o Mycobacterium tuberculosis.
Fig. 2. Modelos de elongación/división celular en diferentes bacterias. (1 stage). Crecimiento a nivel del septo de división en bacterias cocoides que carecen de MreB como Streptococcus pneumoniae. (2 stages). Crecimiento polar en baterias bacilares que carecen de MreB como C. glutamicum o Mycobacterium tuberculosis. (3 stages) Crecimiento dirigido por MreB a lo largo de la pared lateral de B. subtilis o E. coli. El color amarillo muestra los sitios de síntesis de nuevo peptidoglucano y el color rojo muestra el sitio donde se inserta el nuevo peptidoglucano. Tomada de Cabeen & Jacobs-Warner (2007).
Bacterial cell growth and cell division are highly complicated and diversified biological processes. In most rod-shaped bacteria, actin-like MreB homologues produce helicoidal structures along the cell that support elongation of the lateral cell wall. An exception to this rule is peptidoglycan synthesis in the rod-shaped actinomycete Corynebacterium glutamicum, which is MreB-independent. Instead, during cell elongation this bacterium synthesizes new cell-wall material at the cell poles whereas the lateral wall remains inert. Thus, the strategy employed by C. glutamicum to acquire a rod-shaped morphology is completely different from that of Escherichia coli or Bacillus subtilis. Cell division in C. glutamicum also differs profoundly by the apparent absence in its genome of homologues of spatial or temporal regulators of cell division, and its cell division apparatus seems to be simpler than those of other bacteria. Here we review recent advances in our knowledge of the C. glutamicum cell cycle in order to further understand this very different model of rod-shape acquisition.
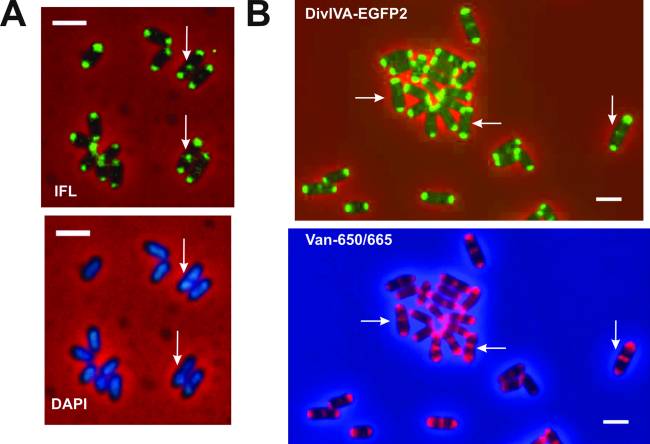
Fig. X. DivIVA se posiciona en la mitad de la célula en un periodo tardío del proceso de división celular de C. glutamicum. (A) Microscopía de inmunofluorescence (IFL) usando anticuerpos anti-DivIVA y tinción DAPI para tenir losnucleoides de C. glutamicum R31. Las flechas muestran que DivIVA se localiza en el septo despues de la segregación de los nucleoides. Barra de tamaño, 1 μm. (B) Microscopía de fluorescencia (EGFP2) y tinción Van-650/665 de C. glutamicum R33, portando una copia cromosomal extra de divIVACg–egfp2 bajo el control del promotor Pdiv (Pdiv-divIVACg–egfp2). Las flechas muestran las bacterias en las que la biosíntesis de nuevo peptidoglucano (visualizado con la tinción Van-650/660) precede a la localization de DivIVA-EGFP2 (fluorescencia verde, EGFP2) en el septo. Barra de tamaño, 1 μm.